DNA-Polymerase-Definition
DNA-Polymerasen sind Enzyme, die die Synthese von DNA-Molekülen aus Desoxyribonukleotiden katalysieren. DNA-Polymerasen spielen eine Schlüsselrolle bei der DNA-Replikation und ermöglichen die Weitergabe genetischer Informationen an Tochterzellen von Generation zu Generation.
Herausgegeben von Christina Swords, Ph.D.
Biochemische Aspekte von DNA-Polymerasen
Polymeraseaktivität
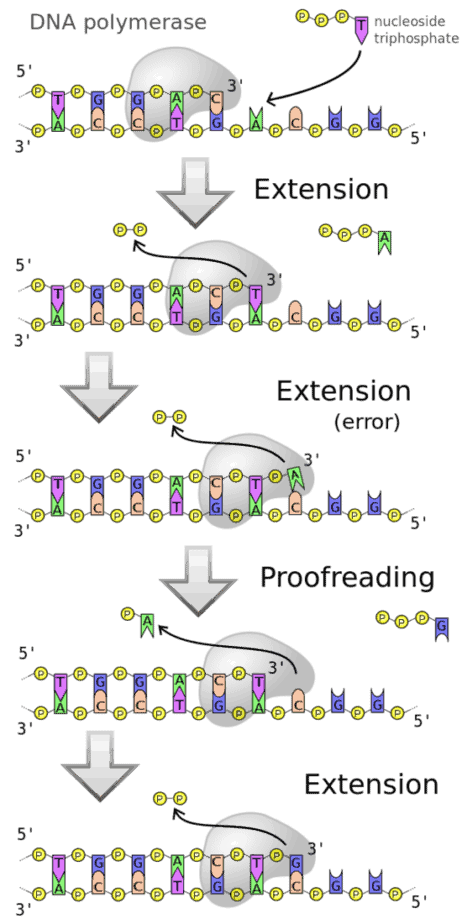
Die Polymerase ermöglicht die chemische Verknüpfung einzelner Moleküle (Monomere) zu einer Kette (Polymer). Im Fall der DNA-Polymerase ist das gebildete Polymer Desoxyribonukleinsäure (DNA). Die Monomere sind Desoxyribonukleotide, genauer gesagt Desoxynukleosidtriphosphate (dNTPs). Die DNA-abhängige DNA-Polymerase verwendet immer einen bereits vorhandenen DNA-Einzelstrang als Matrize für die Synthese eines neuen komplementären Strangs, dessen Nukleotidsequenz somit durch die Matrize bestimmt wird. Diese Erhaltung der DNA-Sequenz ist entscheidend für die Fähigkeit der DNA-Polymerase, die in der DNA kodierte genetische Information zu kopieren. Das korrekte Kopieren der Matrize wird durch komplementäre Basenpaarung der eingebauten Nukleotidbasen mit den Basen der DNA-Matrize erreicht, vermittelt durch Wasserstoffbrücken. Die Synthese des neuen DNA-Strangs erfolgt vom 5′- bis zum 3′-Ende. Chemisch findet ein nukleophiler Angriff der terminalen 3′-Hydroxygruppe des DNA-Strangs auf das α-Phosphat des dNTP statt, wobei Pyrophosphat freigesetzt wird. Dieser Schritt wird durch die Polymerase katalysiert.
Im Gegensatz zu RNA-Polymerasen (produziert RNA, die zur Synthese von Proteinen aus Aminosäuren verwendet wird) kann die Synthese des komplementären DNA-Strangs in DNA-Polymerasen nur stattfinden, wenn der Polymerase ein freies 3′-Hydroxy-Ende zur Verfügung steht. Das erste Nukleotid wird dann an dieses Ende gebunden. Bei der Polymerasekettenreaktion (PCR) wird ein DNA-Einzelstrang (Primer) mit einer Länge von etwa 15 bis 20 Nukleotiden als Ausgangspunkt der Reaktion verwendet. Die Enzyme benötigen üblicherweise Magnesiumionen als Cofaktor.
Die Katalyse der Bildung der Diesterbindung ist funktionell analog zur entsprechenden Reaktion von RNA-Polymerasen. Das letzte bereits synthetisierte Nukleotid des Abschnitts und das hinzuzufügende Nukleotid sind an eines von jeweils zwei Magnesiumionen im katalytischen Zentrum der Polymerasedomäne koordiniert. Die erste Phosphatgruppe des hinzuzufügenden Nukleotids ist an beide Magnesiumionen koordiniert. Die räumliche Position ermöglicht es der Hydroxygruppe des vorhergehenden Nukleotids, die Phosphatgruppe des hinzuzufügenden Nukleotids anzugreifen. Dabei wird ein Pyrophosphatrest abgespalten.
Exonukleaseaktivität
Viele Polymerasen haben auch andere Enzymfunktionen. In Gegenwart geringer Konzentrationen von dNTPs überwiegt die 3 ‚→ 5‘-Exonukleaseaktivität zur Nukleotidentfernung. Einige Polymerasen haben auch eine 5 ‚→ 3‘ Exonukleaseaktivität. Um sicherzustellen, dass beim Lesen der DNA-Matrize keine Fehler auftreten, haben sie diese Korrekturlesefunktion, dh sie können die Insertion eines ungeeigneten Nukleotids nachweisen und es dann mittels der Exonukleaseaktivität aus der DNA entfernen. Dies ermöglicht den Abbau eines vorhandenen DNA- oder RNA-Strangs, der bereits mit dem Matrizenstrang gepaart ist, während ein neuer Strang gebildet wird. Dies führt zu einem Austausch des alten Strangs gegen einen neuen Strang. Diese Exonukleaseaktivität wird durch das Nick-Translationsverfahren ausgenutzt.
Verschiedene DNA-Polymerasen
In Bakterien wie Escherichia coli gibt es drei verschiedene DNA-abhängige DNA-Polymerasen. Eine davon, die DNA-Polymerase I (Pol I), wurde 1955 von Arthur Kornberg isoliert und war die erste Polymerase, die jemals entdeckt wurde. Dies ist jedoch nicht die wichtigste Polymerase für die Replikation in E. coli, da sie nur etwa 20 Syntheseschritte katalysiert (dh nur eine geringe Verarbeitungsleistung aufweist). Aufgrund seiner 5’→ 3′-Exonukleaseaktivität ist es jedoch für den Primerabbau während der Replikation verantwortlich. DNA-Polymerase II und DNA-Polymerase III, die beiden anderen DNA-Polymerasen in E. coli, wurden nur 15 Jahre nach der Entdeckung der DNA-Polymerase I isoliert, nachdem sich E. coli-Mutanten mit einem Defekt im Polymerase I-Gen dennoch als kompetent erwiesen hatten Reproduzieren. Diese Mutanten waren jedoch besonders anfällig für UV-Strahlung und alkylierende Substanzen, weshalb angenommen wird, dass die DNA-Polymerase I hauptsächlich Reparaturaufgaben ausführt. Die Polymerase III, die die eigentliche Replikation in E. coli durchführt, besteht aus insgesamt sieben Untereinheiten und kommt nur in sehr wenigen Kopien pro Bakterienzelle vor.
Eukaryotische DNA-Polymerasen, einschließlich menschlicher DNA-Polymerasen, werden in die folgenden Familien eingeteilt:
- Familie A: DNA-Polymerasen γ, θ und ν
- Familie B: DNA-Polymerasen α, δ, ε und ζ
- Familie X: DNA-Polymerasen β, λ, σ und μ
- Familie Y: DNA-Polymerasen η, ι und κ
Die Polymerase γ kommt nur in Mitochondrien vor.
Bei Säugetieren kommen nur fünf Typen vor: α, β, γ, δ und ε. Es wird angenommen, dass die für die Replikation entscheidenden Polymerasen δ und ε durch eine hohe Verarbeitungsleistung und Korrekturlesefunktion gekennzeichnet sind. Im Gegensatz dazu zeigen die Polymerasen α und β nur eine geringe Verarbeitungsleistung und keine Korrekturlesefunktion.
Darüber hinaus gibt es RNA-abhängige DNA-Polymerasen, die RNA als Matrize verwenden und dNTPs daran binden. Diese werden als reverse Transkriptasen bezeichnet, zu denen auch die Telomerase gehört. Die einzige bekannte unabhängige DNA-Polymerase ist die terminale Desoxyribonukleotidyltransferase.
In Archaebakterien gibt es temperaturstabile Typen, die auch für die PCR verwendet werden.
Biologische Bedeutung
DNA-Polymerasen sind für die DNA-Replikation von zentraler Bedeutung. Sie ermöglichen das getreue Kopieren genetischer Informationen in Form von DNA und sind damit ein entscheidender Schritt bei der Reproduktion und Fortpflanzung lebender Organismen. Die Enzyme spielen auch eine wichtige Rolle bei Prozessen, die mit der DNA-Reparatur verbunden sind.
Biotechnologische Bedeutung
Im Labor werden DNA-Polymerasen häufig für die Polymerasekettenreaktion und verwandte Methoden (z. B. RT-PCR, qPCR) zur Nick-Translation, zum zufälligen Priming und zur DNA-Sequenzierung verwendet. Eine große Anzahl verschiedener thermostabiler Typen (zB Taq-Polymerase aus Thermus aquaticus ) verwendet werden, von denen einige durch Protein-Engineering modifiziert werden. Thermostabile DNA-Polymerasen archaischen Ursprungs wie Pfu-Polymerase liefern neben der Hochtemperaturstabilität auch Korrekturlesen, da die PCR zu keinen Veränderungen der produzierten DNA führen sollte. Weiterhin werden strangverdrängende DNA-Polymerasen wie φ29-DNA-Polymerase in verschiedenen Verfahren zur isothermen DNA-Amplifikation bei Raumtemperatur verwendet. Der Vorläufer der heute verwendeten DNA-Polymerasen war die T4-DNA-Polymerase.
Hat dir dieser Blog-Beitrag gefallen? Sie können finden Weitere Beiträge zu DNA, Genetik und Sequenzierung finden Sie hier !
Sie könnten an folgenden verwandten Blog-Posts interessiert sein:
Bevor Sie abreisen, lesen Sie unsere Sequenzierung des gesamten Genoms!
