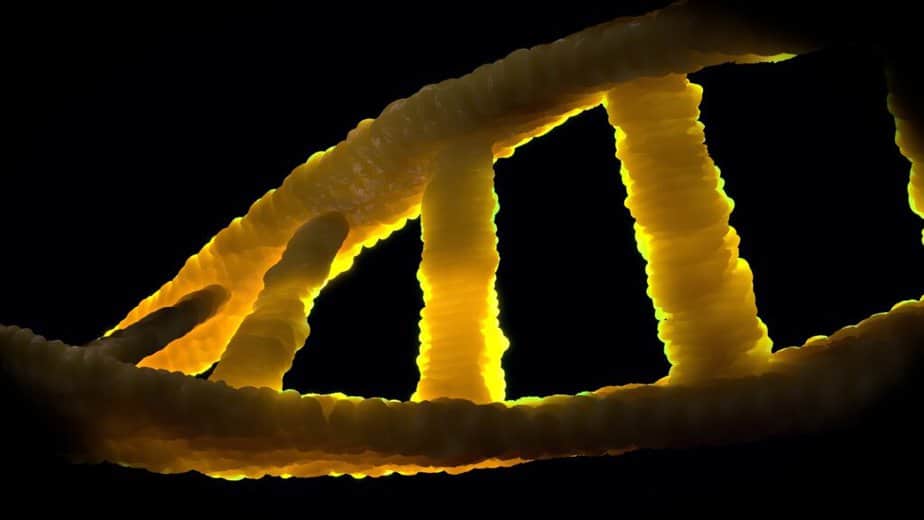
Einführung in die DNA
Desoxyribonukleinsäure ist eine Nukleinsäure, die ein Polynukleotid ist, das aus einer Kette vieler Nukleotide besteht. Das in den Chromosomen befindliche Biomolekül ist der Träger der genetischen Information in allen lebenden Organismen und in vielen Viren (DNA-Viren, Pararetroviren), dh der materiellen Basis der Gene.
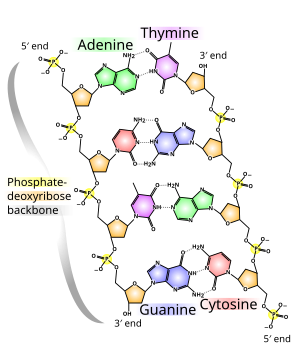
Im Normalzustand ist die DNA in Form einer Doppelhelix aufgebaut. Seine Bausteine sind vier verschiedene Nukleotide, die jeweils aus einem Phosphatrest, der Zuckerdesoxyribose und einer von vier organischen Basen (Adenin, Thymin, Guanin und Cytosin, oft als A, T, G und C abgekürzt) bestehen.
Die Gene in der DNA enthalten die Informationen für die Produktion von Ribonukleinsäuren. In Protein-kodierenden Genen ist dies eine wichtige RNA-Gruppe, mRNA. Es enthält wiederum die Informationen für den Aufbau von Proteinen, die für die biologische Entwicklung eines Lebewesens und den Stoffwechsel in der Zelle notwendig sind. Die Sequenz der Basen bestimmt hier die Sequenz der Aminosäuren des jeweiligen Proteins: Der genetische Code codiert eine spezifische Aminosäure mit jeweils drei benachbarten Basen.
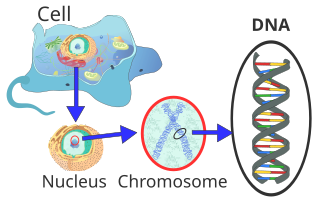
In den Zellen von Eukaryoten, zu denen auch Pflanzen, Tiere und Pilze gehören, ist der Großteil der DNA im Zellkern (lateinischer Kern, daher Kern-DNA oder nDNA) als Chromosomen organisiert. Ein kleiner Teil befindet sich in den Mitochondrien, den „Kraftwerken“ der Zellen, und wird dementsprechend als mitochondriale DNA (mtDNA) bezeichnet. Pflanzen und Algen haben auch DNA in photosynthetischen Organellen, den Chloroplasten oder Plastiden (cpDNA). In Bakterien und Archaeen – den Prokaryoten ohne Zellkern – befindet sich die DNA im Zytoplasma. Einige Viren, sogenannte RNA-Viren, speichern ihre genetische Information in RNA anstelle von DNA.
Herausgegeben von Christina Swords, Ph.D.
Struktur und Organisation der DNA
Bausteine
Desoxyribonukleinsäure ist ein langkettiges Molekül (Polymer), das aus vielen Bausteinen besteht, die als Desoxyribonukleotide oder kurz Nukleotide bezeichnet werden. Jedes Nukleotid besteht aus drei Komponenten: Phosphorsäure oder Phosphat, Zuckerdesoxyribose und einer heterocyclischen Nukleobase oder Base. Die Desoxyribose- und Phosphorsäure-Untereinheiten sind für jedes Nukleotid gleich. Sie bilden das Rückgrat des Moleküls. Einheiten von Base und Zucker (ohne Phosphat) werden als Nukleoside bezeichnet.
Die Phosphatreste sind aufgrund ihrer negativen Ladung hydrophil; Sie geben DNA in wässriger Lösung eine insgesamt negative Ladung. Da diese in Wasser gelöste negativ geladene DNA keine weiteren Protonen abgeben kann, handelt es sich streng genommen nicht (nicht mehr) um eine Säure. Der Begriff Desoxyribonukleinsäure bezieht sich auf einen ungeladenen Zustand, in dem Protonen an die Phosphatreste gebunden sind.
Die Base kann ein Purin sein, nämlich Adenin (A) oder Guanin (G), oder ein Pyrimidin, nämlich Thymin (T) oder Cytosin (C). Da sich die vier verschiedenen Nukleotide nur in ihrer Basis unterscheiden, werden die Abkürzungen A, G, T und C auch für die entsprechenden Nukleotide verwendet.
Die fünf Kohlenstoffatome einer Desoxyribose sind von 1 ‚bis 5′ nummeriert. Die Basis ist an das 1′-Ende dieses Zuckers gebunden. Der Phosphatrest ist an das 5′-Ende gebunden. Genau genommen ist Desoxyribose 2-Desoxyribose; Der Name kommt von der Tatsache, dass im Vergleich zu einem Ribosemolekül eine alkoholische Hydroxygruppe (OH-Gruppe) an der 2‘-Position fehlt (dh durch ein Wasserstoffatom ersetzt wurde).
An der 3′-Position befindet sich eine OH-Gruppe, die die Desoxyribose über eine sogenannte Phosphodiesterbindung mit dem 5′-Kohlenstoffatom des Zuckers des nächsten Nukleotids verbindet. Somit hat jeder sogenannte Einzelstrang zwei verschiedene Enden: ein 5′- und ein 3′-Ende. DNA-Polymerasen, die DNA-Stränge in der lebenden Welt synthetisieren, können nur am 3′-Ende neue Nukleotide an die OH-Gruppe binden, nicht jedoch am 5′-Ende. Der Einzelstrang wächst daher immer von 5 ‚auf 3‘. Als neuer Baustein wird ein Nukleosidtriphosphat (mit drei Phosphatresten) geliefert, von dem zwei Phosphate in Form von Pyrophosphat abgespalten werden. Der verbleibende Phosphatrest jedes neu hinzugefügten Nukleotids ist durch Abspalten von Wasser mit der OH-Gruppe am 3′-Ende des letzten im Strang vorhandenen Nukleotids verbunden. Die Sequenz der Basen im Strang codiert die genetische Information.
Die Doppelhelix
DNA tritt normalerweise als helikale Doppelhelix in einer Konformation auf, die als B-DNA bezeichnet wird. Zwei der oben beschriebenen Einzelstränge sind in entgegengesetzten Richtungen miteinander verbunden: An jedem Ende der Doppelhelix hat einer der beiden Einzelstränge sein 3′-Ende, der andere sein 5′-Ende. Aufgrund des Nebeneinander liegen sich zwei spezifische Basen in der Mitte der Doppelhelix immer gegenüber, sie sind „gepaart“. Die Doppelhelix wird hauptsächlich durch Stapelwechselwirkungen zwischen aufeinanderfolgenden Basen desselben Strangs stabilisiert (und nicht, wie oft behauptet, durch Wasserstoffbrücken zwischen den Strängen).
Adenin und Thymin sind immer gepaart und bilden zwei Wasserstoffbrücken oder Cytosin mit Guanin, die durch drei Wasserstoffbrücken verbunden sind. Eine Brücke wird zwischen den molekularen Positionen 1═1 und 6═6 und bei Guanin-Cytosin-Paarungen zusätzlich zwischen 2═2 gebildet. Da immer die gleichen Basen gepaart sind, kann die Sequenz der Basen in einem Strang verwendet werden, um die des anderen Strangs abzuleiten. Die Sequenzen sind komplementär. Die Wasserstoffbrückenbindungen sind fast ausschließlich für die Spezifität der Paarung verantwortlich, nicht jedoch für die Stabilität der Doppelhelix.
Da ein Purin immer mit einem Pyrimidin kombiniert wird, ist der Abstand zwischen den Strängen überall gleich, es entsteht eine regelmäßige Struktur. Die gesamte Helix hat einen Durchmesser von etwa 2 nm und windet sich mit jedem Zuckermolekül um 0,34 nm weiter.
Die Ebenen der Zuckermoleküle stehen in einem Winkel von 36 ° zueinander, und daher wird nach 10 Basen (360 °) und 3,4 nm eine vollständige Rotation erreicht. DNA-Moleküle können sehr groß werden. Beispielsweise enthält das größte menschliche Chromosom 247 Millionen Basenpaare.
Wenn die beiden Einzelstränge miteinander verdrillt werden, bleiben seitliche Lücken bestehen, so dass sich die Basen hier direkt auf der Oberfläche befinden. Es gibt zwei dieser Rillen, die sich um die Doppelhelix wickeln (siehe Abbildungen und Animationen am Anfang des Artikels). Die „große Furche“ ist 2,2 nm breit, die „kleine Furche“ nur 1,2 nm.
Dementsprechend sind die Basen in der großen Furche leichter zugänglich. Proteine, die sequenzspezifisch an die DNA binden, wie z. B. Transkriptionsfaktoren, binden daher normalerweise an die große Furche.
Einige DNA-Farbstoffe, beispielsweise DAPI, binden auch an eine Furche.
Die kumulative Bindungsenergie zwischen den beiden Einzelsträngen hält sie zusammen. Kovalente Bindungen sind hier nicht vorhanden, was bedeutet, dass die DNA-Doppelhelix nicht aus einem Molekül besteht, sondern aus zwei. Dadurch können die beiden Stränge in biologischen Prozessen vorübergehend getrennt werden.
Neben der soeben beschriebenen B-DNA gibt es auch A-DNA und eine linkshändige sogenannte Z-DNA, die erstmals 1979 von Alexander Rich und seinen Kollegen am MIT untersucht wurde. Dies tritt insbesondere in GC-reichen Abschnitten auf. Erst 2005 wurde eine Kristallstruktur beschrieben, die Z-DNA direkt in einer Verbindung mit B-DNA zeigt und somit Hinweise auf die biologische Aktivität von Z-DNA liefert. Die folgende Tabelle und die nebenstehende Abbildung zeigen die Unterschiede zwischen den drei Formen im direkten Vergleich.
Die Basisstapel sind nicht genau wie Bücher parallel zueinander, sondern bilden Keile, die die Helix in die eine oder andere Richtung neigen. Der größte Keil wird von Adenosinen gebildet, die mit Thymidinen des anderen Strangs gepaart sind. Folglich bildet eine Reihe von AT-Paaren einen Bogen in der Helix. Wenn solche Reihen in kurzen Abständen aufeinander folgen, nimmt das DNA-Molekül eine gekrümmte oder gebogene Struktur an, die stabil ist. Dies wird auch als sequenzinduzierte Beugung bezeichnet, da die Beugung auch durch Proteine induziert werden kann (sogenannte proteininduzierte Beugung). Sequenzinduzierte Beugung findet sich häufig an wichtigen Stellen im Genom.
Chromatin und Chromosomen
Menschliche Chromosomen in der späten Metaphase der mitotischen Zellkernteilung: Jedes Chromosom zeigt zwei Chromatiden, die in der Anaphase getrennt und in zwei Zellkerne aufgeteilt sind.
In der eukaryotischen Zelle ist die DNA in Form von Chromatinfäden organisiert, die als Chromosomen bezeichnet werden und sich im Zellkern befinden. Ein einzelnes Chromosom enthält einen langen, kontinuierlichen DNA-Doppelstrang (in einem Chromatid) von der Anaphase bis zum Beginn der S-Phase. Am Ende der S-Phase besteht das Chromosom aus zwei identischen DNA-Strängen (in zwei Chromatiden).
Da ein solcher DNA-Faden mehrere Zentimeter lang sein kann, ein Zellkern jedoch nur wenige Mikrometer im Durchmesser hat, muss die DNA zusätzlich komprimiert oder „gepackt“ werden. Bei Eukaryoten geschieht dies mit sogenannten Chromatinproteinen, von denen die basischen Histone besonders hervorzuheben sind. Sie bilden die Nukleosomen, um die die DNA auf der niedrigsten Verpackungsebene gewickelt ist. Während der Kernteilung (Mitose) wird jedes Chromosom zu seiner maximal kompakten Form kondensiert. Dadurch können sie während der Metaphase besonders gut unter dem Lichtmikroskop identifiziert werden.
Bakterielle und virale DNA
In prokaryotischen Zellen liegt der Großteil der doppelsträngigen DNA in den bisher dokumentierten Fällen nicht als lineare Stränge mit jeweils einem Anfang und einem Ende vor, sondern als kreisförmige Moleküle – jedes Molekül (dh jeder DNA-Strang) schließt einen Kreis mit seiner 3 ‚und 5‘ enden. Diese beiden zirkulären, geschlossenen DNA-Moleküle werden je nach Länge der Sequenz als bakterielles Chromosom oder Plasmid bezeichnet. In Bakterien befinden sie sich ebenfalls nicht in einem Zellkern, sondern sind im Plasma frei vorhanden. Die prokaryotische DNA wird mit Hilfe von Enzymen (z. B. Topoisomerasen und Gyrasen), die einem klingelnden Telefonkabel ähneln, zu einfachen „Superspulen“ aufgewickelt. Indem die Helices immer noch um sich selbst gedreht werden, wird der für die genetische Information erforderliche Platz reduziert. In den Bakterien stellen Topoisomerasen sicher, dass der verdrillte Doppelstrang an einer gewünschten Stelle gerungen wird, indem sie die DNA ständig schneiden und wieder verbinden (Voraussetzung für die DNA-Transkription und DNA-Replikation). Viren enthalten je nach Typ entweder DNA oder RNA als genetische Information. Sowohl in DNA- als auch in RNA-Viren ist die Nukleinsäure durch eine Proteinhülle geschützt.
Chemische und physikalische Eigenschaften der DNA-Doppelhelixstruktur
Bei neutralem pH-Wert ist DNA ein negativ geladenes Molekül, wobei sich die negativen Ladungen an den Phosphatgruppen im Rückgrat der Stränge befinden. Obwohl zwei der drei sauren OH-Gruppen der Phosphate mit den jeweiligen benachbarten Desoxyribosen verestert sind, ist die dritte noch vorhanden und setzt bei neutralem pH ein Proton frei, das die negative Ladung verursacht. Diese Eigenschaft wird bei der Agarosegelelektrophorese verwendet, um verschiedene DNA-Stränge entsprechend ihrer Länge zu trennen. Einige physikalische Eigenschaften wie die freie Energie und der Schmelzpunkt der DNA stehen in direktem Zusammenhang mit dem GC-Gehalt, dh sie sind sequenzabhängig.
DNA-Stapel-Wechselwirkungen
Zwei Hauptfaktoren sind für die Stabilität der Doppelhelix verantwortlich: Basenpaarung zwischen komplementären Basen und Stapelwechselwirkungen zwischen aufeinanderfolgenden Basen.
Entgegen der ursprünglichen Annahme ist der Energiegewinn durch Wasserstoffbrückenbindung vernachlässigbar, da die Basen mit dem umgebenden Wasser ähnlich gute Wasserstoffbrückenbindungen eingehen können. Die Wasserstoffbrücken eines GC-Basenpaars tragen nur minimal zur Stabilität der Doppelhelix bei, während die eines AT-Basenpaars sogar destabilisierend wirken. Stapelwechselwirkungen wirken sich dagegen nur auf die Doppelhelix zwischen aufeinanderfolgenden Basenpaaren aus: Zwischen den aromatischen Ringsystemen der heterocyclischen Basen entsteht eine dipolinduzierte Dipolwechselwirkung, die energetisch günstig ist. Somit ist die Bildung des ersten Basenpaars aufgrund des geringen Energiegewinns und -verlusts ziemlich ungünstig, aber die Dehnung (Verlängerung) der Helix ist energetisch günstig, da das Stapeln des Basenpaars unter Energiegewinn stattfindet.
Die Stapelwechselwirkungen sind jedoch sequenzabhängig und energetisch am günstigsten für gestapelte GC-GC, weniger günstig für gestapelte AT-AT. Die Unterschiede in den Stapelwechselwirkungen erklären hauptsächlich, warum GC-reiche DNA-Schnitte thermodynamisch stabiler sind als AT-reiche Schnitte, während Wasserstoffbrücken eine untergeordnete Rolle spielen.
DNA-Schmelzpunkt
Der Schmelzpunkt der DNA ist die Temperatur, bei der die Bindungskräfte zwischen den beiden Einzelsträngen überwunden werden und sich voneinander trennen. Dies wird auch Denaturierung genannt.
Solange die DNA in einem kooperativen Übergang (der in einem engen Temperaturbereich stattfindet) denaturiert wird, ist der Schmelzpunkt die Temperatur, bei der die Hälfte der Doppelstränge zu Einzelsträngen denaturiert wird. Aus dieser Definition werden die korrekten Begriffe „Mittelpunkt der Übergangstemperatur“ Tm abgeleitet.
Der Schmelzpunkt hängt von der jeweiligen Basensequenz in der Helix ab. Sie nimmt zu, wenn die Helix mehr GC-Basenpaare enthält, da diese entropisch günstiger sind als AT-Basenpaare. Dies ist nicht so sehr auf die unterschiedliche Anzahl von Wasserstoffbrücken zurückzuführen, die von den beiden Paaren gebildet werden, sondern vielmehr auf die unterschiedlichen Stapelwechselwirkungen. Die Stapelnergie von zwei Basenpaaren ist viel kleiner, wenn eines der beiden Paare ein AT-Basenpaar ist. GC-Stapel hingegen sind energetisch günstiger und stabilisieren die Doppelhelix stärker. Das Verhältnis der GC-Basenpaare zur Gesamtzahl aller Basenpaare ergibt sich aus dem GC-Gehalt.
Da einzelsträngige DNA UV-Licht etwa 40 Prozent stärker absorbiert als doppelsträngige DNA, kann die Übergangstemperatur in einem Photometer leicht bestimmt werden.
Wenn die Temperatur der Lösung unter Tm fällt, können sich die einzelnen Stränge wieder verbinden. Dieser Prozess wird als Renaturierung oder Hybridisierung bezeichnet. Das Zusammenspiel von De- und Renaturierung wird in vielen biotechnologischen Prozessen genutzt, beispielsweise bei der Polymerasekettenreaktion (PCR), Southern Blots und der In-situ-Hybridisierung.
Hat dir dieser Blog-Beitrag gefallen? Sie können finden Weitere Beiträge zu DNA, Genetik und Sequenzierung finden Sie hier !
Sie könnten an folgenden verwandten Blog-Posts interessiert sein:
Bevor Sie abreisen, lesen Sie unsere Sequenzierung des gesamten Genoms!