Introdução ao DNA
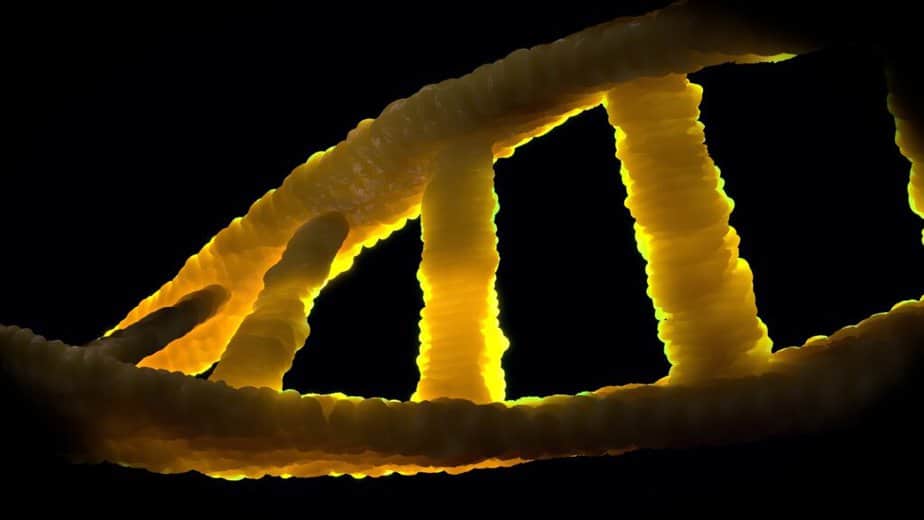
O ácido desoxirribonucleico é um ácido nucleico que é um polinucleotídeo composto por uma cadeia de muitos nucleotídeos. A biomolécula localizada nos cromossomos é a portadora da informação genética em todos os organismos vivos e em muitos vírus (vírus de DNA, pararetrovírus), ou seja, a base material dos genes.
No estado normal, o DNA é construído na forma de uma dupla hélice. Seus blocos de construção são quatro nucleotídeos diferentes, cada um consistindo de um resíduo de fosfato, a desoxirribose de açúcar e uma de quatro bases orgânicas (adenina, timina, guanina e citosina, freqüentemente abreviados como A, T, G e C).
Os genes do DNA contêm as informações para a produção de ácidos ribonucléicos. Em genes que codificam proteínas, este é um importante grupo de RNA, mRNA. Por sua vez, contém as informações para a construção das proteínas, necessárias ao desenvolvimento biológico de um ser vivo e ao metabolismo na célula. A sequência de bases aqui determina a sequência de aminoácidos da respectiva proteína: O código genético codifica um aminoácido específico com três bases adjacentes cada.
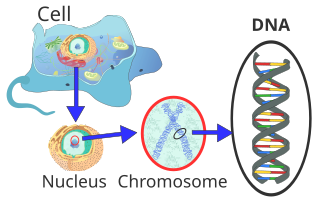
Nas células dos eucariotos, que também incluem plantas, animais e fungos, a maior parte do DNA do núcleo da célula (núcleo latino, portanto DNA nuclear ou nDNA) é organizado como cromossomos. Uma pequena parte está localizada nas mitocôndrias, as “estações de energia” das células, e é chamada de DNA mitocondrial (mtDNA). Plantas e algas também possuem DNA em organelas fotossintéticas, os cloroplastos ou plastídios (cpDNA). Em bactérias e arqueas – os procariontes que não possuem núcleo celular – o DNA está localizado no citoplasma. Alguns vírus, os chamados vírus de RNA, armazenam sua informação genética no RNA em vez de no DNA.
Editado por Christina Swords, Ph.D.
Estrutura e Organização do DNA
Blocos de construção
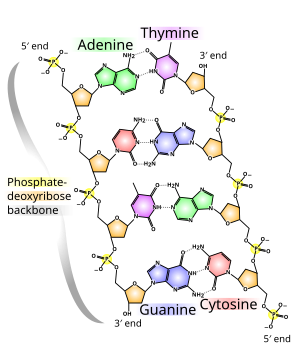
O ácido desoxirribonucleico é uma molécula de cadeia longa (polímero) composta de muitos blocos de construção chamados desoxirribonucleotídeos ou, para abreviar, nucleotídeos. Cada nucleotídeo tem três componentes: ácido fosfórico ou fosfato, o açúcar desoxirribose e uma nucleobase ou base heterocíclica. As subunidades de desoxirribose e ácido fosfórico são iguais para cada nucleotídeo. Eles formam a espinha dorsal da molécula. As unidades de base e açúcar (sem fosfato) são chamadas de nucleosídeos.
Os resíduos de fosfato são hidrofílicos devido à sua carga negativa; eles dão ao DNA em solução aquosa uma carga geral negativa. Uma vez que esse DNA com carga negativa dissolvido em água não pode liberar mais prótons, ele não é (não mais) um ácido estritamente falando. O termo ácido desoxirribonucléico refere-se a um estado sem carga no qual os prótons estão ligados aos resíduos de fosfato.
A base pode ser uma purina, nomeadamente adenina (A) ou guanina (G), ou uma pirimidina, nomeadamente timina (T) ou citosina (C). Uma vez que os quatro nucleotídeos diferentes diferem apenas em sua base, as abreviações A, G, T e C também são usadas para os nucleotídeos correspondentes.
Os cinco átomos de carbono de uma desoxirribose são numerados de 1 ‘a 5’. A base está ligada à extremidade 1 ‘deste açúcar. O resíduo de fosfato é ligado à extremidade 5 ‘. Estritamente falando, a desoxirribose é 2-desoxirribose; o nome vem do fato de que, em comparação com uma molécula de ribose, um grupo hidroxila alcoólica (grupo OH) está faltando na posição 2 ‘(ou seja, foi substituído por um átomo de hidrogênio).
Na posição 3 ‘há um grupo OH que liga a desoxirribose ao átomo de carbono 5’ do açúcar do próximo nucleotídeo por meio de uma chamada ligação fosfodiéster. Assim, cada uma das chamadas fitas simples tem duas extremidades diferentes: uma extremidade 5 ‘e uma extremidade 3’. As polimerases de DNA que sintetizam fitas de DNA no mundo vivo só podem anexar novos nucleotídeos ao grupo OH na extremidade 3 ‘, mas não na extremidade 5’. A fita simples, portanto, sempre cresce de 5 ‘para 3’. Um trifosfato de nucleosídeo (com três resíduos de fosfato) é fornecido como um novo bloco de construção, a partir do qual dois fosfatos são separados na forma de pirofosfato. O resíduo de fosfato restante de cada nucleotídeo recém-adicionado é conectado ao grupo OH na extremidade 3 ‘do último nucleotídeo presente na fita por separação da água. A sequência de bases na fita codifica a informação genética.
A dupla hélice
O DNA normalmente ocorre como uma dupla hélice helicoidal em uma conformação chamada B-DNA. Duas das fitas simples descritas acima estão ligadas uma à outra em direções opostas: em cada extremidade da dupla hélice, uma das duas fitas simples tem sua extremidade 3 ‘, a outra, sua extremidade 5’. Por causa da justaposição, duas bases específicas estão sempre opostas uma à outra no meio da dupla hélice, elas são “emparelhadas”. A dupla hélice é principalmente estabilizada por empilhamento de interações entre bases sucessivas da mesma fita (e não, como frequentemente afirmado, por ligações de hidrogênio entre as fitas).
Adenina e timina estão sempre emparelhadas, formando duas ligações de hidrogênio, ou citosina com guanina, que são conectadas por três pontes de hidrogênio. Uma ponte é formada entre as posições moleculares 1═1 e 6═6 e, no caso de emparelhamentos guanina-citosina, adicionalmente entre 2═2. Uma vez que as mesmas bases estão sempre emparelhadas, a sequência das bases em uma fita pode ser usada para derivar as da outra fita, as sequências são complementares. As ligações de hidrogênio são quase exclusivamente responsáveis pela especificidade do emparelhamento, mas não pela estabilidade da dupla hélice.
Como uma purina está sempre combinada com uma pirimidina, a distância entre os fios é a mesma em todos os lugares, forma-se uma estrutura regular. A hélice inteira tem um diâmetro de cerca de 2 nm e enrola-se mais 0,34 nm com cada molécula de açúcar.
Os planos das moléculas de açúcar formam um ângulo de 36 ° entre si e, portanto, a rotação completa é alcançada após 10 bases (360 °) e 3,4 nm. As moléculas de DNA podem se tornar muito grandes. Por exemplo, o maior cromossomo humano contém 247 milhões de pares de bases.
Quando os dois fios individuais são torcidos juntos, permanecem lacunas laterais, de modo que as bases aqui estão diretamente na superfície. Existem duas dessas ranhuras que envolvem a dupla hélice (veja as ilustrações e animação no início do artigo). O “sulco grande” tem 2,2 nm de largura, o “sulco pequeno” apenas 1,2 nm.
Conseqüentemente, as bases no grande sulco são mais acessíveis. As proteínas que se ligam ao DNA de uma maneira específica para a sequência, como os fatores de transcrição, geralmente se ligam ao sulco grande.
Alguns corantes de DNA, por exemplo DAPI, também se ligam a um sulco.
A energia de ligação cumulativa entre as duas fitas simples os mantém juntos. As ligações covalentes não estão presentes aqui, o que significa que a dupla hélice do DNA não consiste em uma molécula, mas em duas. Isso permite que as duas fitas sejam temporariamente separadas em processos biológicos.
Além do B-DNA que acabamos de descrever, há também um A-DNA e um canhoto, chamado Z-DNA, que foi estudado pela primeira vez em 1979 por Alexander Rich e seus colegas no MIT. Isso ocorre especialmente em seções ricas em GC. Não foi até 2005 que uma estrutura de cristal foi relatada que mostra Z-DNA diretamente em um composto com B-DNA e, portanto, fornece evidências para a atividade biológica de Z-DNA. A tabela a seguir e a figura ao lado mostram as diferenças entre as três formas em comparação direta.
Os empilhamento de base não são exatamente paralelos entre si como livros, mas formam cunhas que inclinam a hélice em uma direção ou outra. A maior cunha é formada por adenosinas emparelhadas com timidinas da outra fita. Conseqüentemente, uma série de pares AT forma um arco na hélice. Quando essas séries se sucedem em intervalos curtos, a molécula de DNA assume uma estrutura curva ou dobrada, que é estável. Isso também é chamado de difração induzida por sequência, uma vez que a difração também pode ser induzida por proteínas (a chamada difração induzida por proteína). A difração induzida por sequência é frequentemente encontrada em locais importantes do genoma.
Cromatina e cromossomos
Cromossomos humanos na metáfase tardia da divisão do núcleo da célula mitótica: Cada cromossomo mostra duas cromátides, que são separadas na anáfase e divididas em dois núcleos celulares.
Na célula eucariótica, o DNA é organizado na forma de fios de cromatina, chamados cromossomos, que estão localizados no núcleo da célula. Um único cromossomo contém uma fita dupla de DNA longa e contínua (em uma cromátide) da anáfase ao início da fase S. No final da fase S, o cromossomo consiste em duas fitas de DNA idênticas (em duas cromátides).
Uma vez que tal filamento de DNA pode ter vários centímetros de comprimento, mas um núcleo celular tem apenas alguns micrômetros de diâmetro, o DNA deve ser adicionalmente comprimido ou “compactado”. Em eucariotos, isso é feito com as chamadas proteínas da cromatina, das quais as histonas básicas são particularmente notáveis. Eles formam os nucleossomos em torno dos quais o DNA é envolvido no nível de embalagem mais baixo. Durante a divisão nuclear (mitose), cada cromossomo é condensado em sua forma compacta máxima. Isso permite que eles sejam identificados particularmente bem sob o microscópio de luz durante a metáfase.
DNA bacteriano e viral
Em células procarióticas, a maioria do DNA de fita dupla nos casos documentados até agora não está presente como fitas lineares com um início e um fim cada, mas como moléculas circulares – cada molécula (ou seja, cada fita de DNA) fecha um círculo com seus 3 final ‘e 5’. Essas duas moléculas de DNA circulares fechadas são chamadas de cromossomo bacteriano ou plasmídeo, dependendo do comprimento da sequência. Nas bactérias, eles também não estão localizados no núcleo da célula, mas estão livremente presentes no plasma. O DNA procariótico é enrolado em “supercoils” simples com a ajuda de enzimas (por exemplo topoisomerases e girases), que se assemelham a um fio telefônico com anel. Ao ainda girar as hélices em torno de si mesmas, o espaço necessário para a informação genética é reduzido. Nas bactérias, as topoisomerases garantem que a fita dupla torcida seja capturada no local desejado, cortando e reunindo o DNA constantemente (pré-requisito para a transcrição e replicação do DNA). Dependendo do tipo, os vírus contêm DNA ou RNA como informação genética. Em ambos os vírus DNA e RNA, o ácido nucleico é protegido por um envelope de proteína.
Propriedades químicas e físicas da estrutura de dupla hélice do DNA
Em pH neutro, o DNA é uma molécula carregada negativamente, com as cargas negativas localizadas nos grupos fosfato na espinha dorsal das fitas. Embora dois dos três grupos OH ácidos dos fosfatos estejam esterificados com as respectivas desoxirriboses vizinhas, o terceiro ainda está presente e libera um próton em pH neutro, que causa a carga negativa. Esta propriedade é usada na eletroforese em gel de agarose para separar diferentes fitas de DNA de acordo com seu comprimento. Algumas propriedades físicas, como a energia livre e o ponto de fusão do DNA, estão diretamente relacionadas ao conteúdo de GC, ou seja, são dependentes da sequência.
Interações de pilha de DNA
Dois fatores principais são responsáveis pela estabilidade da dupla hélice: pareamento de bases entre bases complementares e interações de empilhamento entre bases consecutivas.
Ao contrário da suposição inicial, o ganho de energia por meio das ligações de hidrogênio é insignificante, uma vez que as bases podem formar ligações de hidrogênio igualmente boas com a água circundante. As ligações de hidrogênio de um par de bases GC contribuem apenas minimamente para a estabilidade da dupla hélice, enquanto as de um par de bases AT têm até um efeito desestabilizador. As interações de pilha, por outro lado, só têm efeito na dupla hélice entre pares de bases sucessivos: Entre os sistemas de anéis aromáticos das bases heterocíclicas, forma-se uma interação dipolo induzida por dipolo, que é energeticamente favorável. Assim, a formação do primeiro par de bases é bastante desfavorável devido ao baixo ganho e perda de energia, mas o alongamento (alongamento) da hélice é energeticamente favorável, uma vez que o empilhamento do par de bases ocorre sob ganho de energia.
No entanto, as interações de empilhamento são dependentes da sequência e energeticamente mais favoráveis para GC-GC empilhado, menos favoráveis para AT-AT empilhado. As diferenças nas interações de empilhamento explicam principalmente porque as seções de DNA ricas em GC são termodinamicamente mais estáveis do que as seções ricas em AT, enquanto as ligações de hidrogênio desempenham um papel menor.
Ponto de fusão do DNA
O ponto de fusão do DNA é a temperatura na qual as forças de ligação entre as duas fitas individuais são superadas e elas se separam. Isso também é chamado de desnaturação.
Enquanto o DNA é desnaturado em uma transição cooperativa (que ocorre em uma faixa estreita de temperatura), o ponto de fusão é a temperatura na qual metade das fitas duplas são desnaturadas em fitas simples. Desta definição, os termos corretos “ponto médio da temperatura de transição” Tm são derivados.
O ponto de fusão depende da respectiva sequência de bases na hélice. Ele aumenta se houver mais pares de bases GC na hélice, uma vez que estes são entropicamente mais favoráveis do que pares de bases AT. Isso não se deve tanto ao número diferente de ligações de hidrogênio formadas pelos dois pares, mas sim às diferentes interações de empilhamento. A energia de empilhamento de dois pares de bases é muito menor se um dos dois pares for um par de bases AT. As pilhas de GC, por outro lado, são energeticamente mais favoráveis e estabilizam a dupla hélice com mais força. A proporção dos pares de bases GC para o número total de todos os pares de bases é dada pelo conteúdo GC.
Como o DNA de fita simples absorve luz ultravioleta cerca de 40% mais fortemente do que o DNA de fita dupla, a temperatura de transição pode ser facilmente determinada em um fotômetro.
Se a temperatura da solução cair abaixo de Tm, os fios simples podem se juntar novamente. Este processo é denominado renaturação ou hibridização. A interação entre desnaturação e renaturação é explorada em muitos processos biotecnológicos, por exemplo, na reação em cadeia da polimerase (PCR), Southern blots e hibridização in situ.
Você gostou desta postagem do blog? Você pode encontrar mais post sobre DNA, genética e sequenciamento aqui !
Você pode se interessar pelas seguintes postagens de blog relacionadas:
Antes de sair, confira nosso Sequenciamento do Genoma Completo!