Table des matières
- Qu’est-ce que le séquençage de l’exome entier?
- Effectuer un séquençage d’exome entier
- Qui devrait se faire tester?
- Avantages du séquençage de l’exome entier
- Limitations du séquençage de l’exome entier
- Microarray chromosomique vs séquençage de l’exome entier vs séquençage du génome entier
- Direct aux entreprises de séquençage d’exome complet
Qu’est-ce que le séquençage de l’exome entier?
Le séquençage de l’exome entier (WES) est le séquençage de l’exome, tous les gènes codant pour les protéines du génome. Le séquençage de l’exome entier comprend deux étapes. La première étape consiste à cibler les séquences dans l’exome humain uniquement. En génétique humaine, ces régions cibles représentent environ 60 millions de paires de bases, soit environ 1% du génome humain de référence. La deuxième étape consiste à séquencer l’ADN à l’aide de n’importe quelle plateforme de séquençage d’ADN à haut débit et à analyser les résultats.
Le séquençage de l’exome permet de détecter les changements génétiques qui conduisent à des changements dans les séquences protéiques, qui peuvent à leur tour conduire à des maladies telles que l’athérosclérose , La maladie d’Alzheimer , et d’autres. De nombreuses maladies surviennent dans la région codant pour les protéines du génome et ce processus permet aux utilisateurs d’en savoir plus sur leur prédisposition à la maladie que l’analyse d’un seul gène. Cependant, il est maintenant connu que les variations d’ADN en dehors des exons peuvent affecter l’activité génique et la production de protéines et conduire également à des troubles génétiques. Le séquençage complet de l’exome manquerait ces variations.
Dans le passé, le principal avantage du séquençage de l’exome était la capacité d’effectuer un plus grand criblage de gènes et de détecter les mutations associées à la maladie, telles que les variantes du nombre de copies, d’une manière rentable et consommant moins de temps que le séquençage du génome entier. Cependant, grâce aux progrès de la technologie de séquençage, le séquençage du génome entier est désormais tout aussi rapide et tout aussi abordable. Génomique des nébuleuses offre le séquençage du génome entier 30X le plus abordable à 299 $ ou moins. Nous décodons 100% de votre ADN, pas seulement 1% comme avec le séquençage complet de l’exome, vous offrant la vue la plus complète de votre ADN.
Effectuer un séquençage d’exome entier
Le séquençage de l’exome entier commence par l’isolement et l’enrichissement de l’ADN et se termine par le séquençage et l’analyse de l’ADN.
Stratégies d’isolement et d’enrichissement de l’ADN
L’ADN est isolé à partir d’un échantillon fourni, tel qu’un échantillon de salive ou un prélèvement de joue. La forme la plus courante d’isolement d’ADN est l’extraction au phénol-chloroforme.
Il existe plusieurs stratégies pour enrichir l’ADN à partir d’échantillons. La méthode utilisée dépend souvent de l’échantillon et du résultat souhaité.
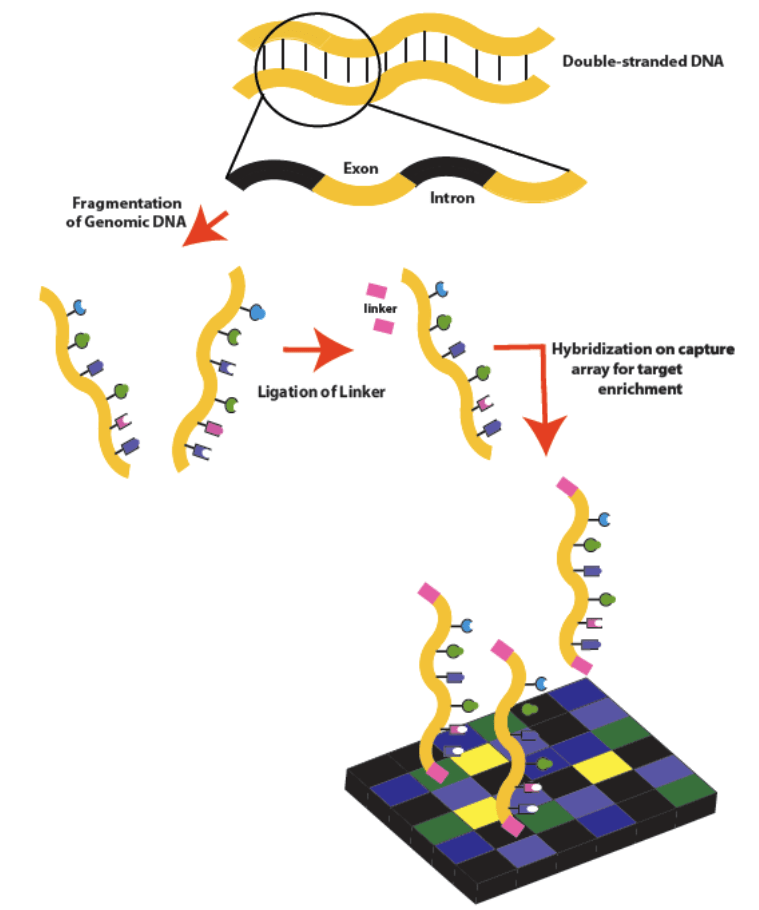
- PCR: La réaction en chaîne par polymérase (PCR) est le moyen le plus courant d’amplifier les fragments d’ADN souhaités pour le séquençage.
- Inversion moléculaire: Cette méthode consiste à fermer la région d’intérêt dans un anneau. Une amorce oligonucléotidique d’ADN simple brin contient une partie centrale avec une séquence universelle avec des sites de restriction, tandis que les extrémités sont complémentaires des deux sections d’ADN génomique entre lesquelles se situe la séquence d’intérêt.
- Hybridation, capture par matrice: on utilise des microréseaux contenant des oligonucléotides simple brin attachés à un substrat avec des séquences du génome capables de couvrir les sites d’intérêt. L’ADN génomique est coupé en fragments, les extrémités des fragments sont émoussées et des amorces universelles sont ajoutées. Après hybridation des fragments avec des sondes sur microréseaux, les fragments non hybrides sont lavés du substrat, et les fragments restants sont ensuite amplifiés par PCR
- Capture en solution: Un ensemble de sondes est synthétisé en solution et fixé sur des billes de streptavidine. Les billes sont placées dans une solution avec de l’ADN génomique fragmenté, où les sondes sont sélectivement hybridées aux sites génomiques souhaités, après quoi les billes avec les fragments d’intérêt sont précipitées et lavées. Les sections sont ensuite séquencées.
Séquençage ADN
Il y a beaucoup de Séquençage de nouvelle génération (NGS) plates-formes technologiques disponibles. Certains incluent le séquenceur Roche 454 et les systèmes Life Technologies SOLiD, le Life Technologies Ion Torrent et Illumina Genome Analyzer II (anciennement) et les instruments des séries Illumina MiSeq, HiSeq et NovaSeq ultérieurs. ‘Lecture courte’ Les systèmes NGS sont idéaux pour analyser de nombreux segments relativement courts de séquence d’ADN, tels que trouvés dans l’exome.
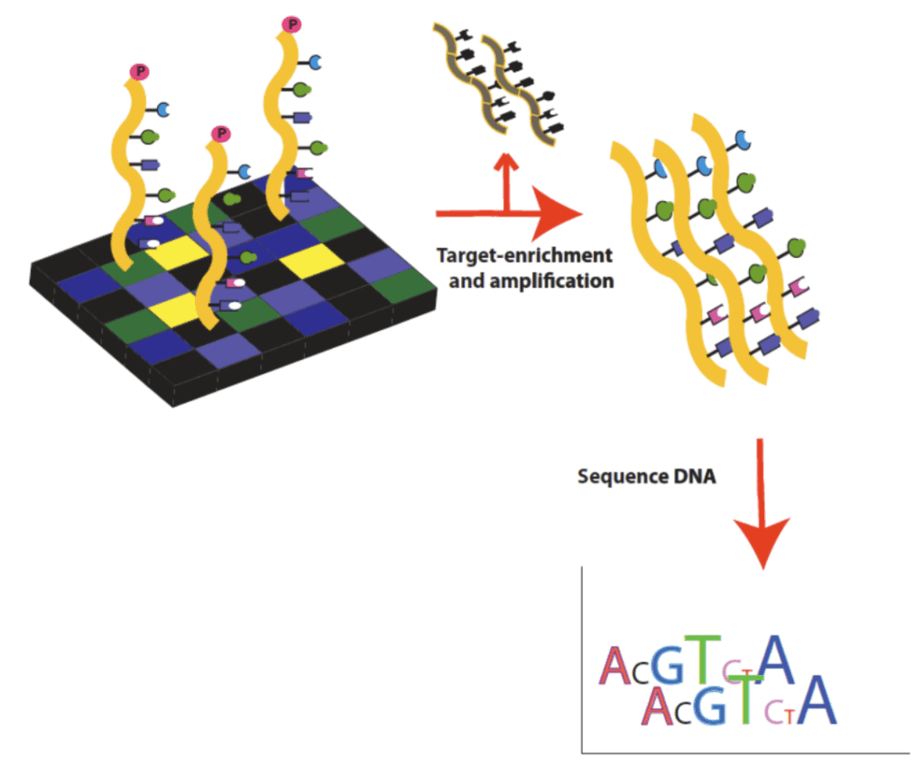
L’analyse des données
Même le séquençage à seulement 1% du génome humain génère beaucoup de données de séquençage d’exome et nécessite une analyse de données lourde, pour laquelle il existe de nombreux programmes pour aligner et assembler les lectures de séquence. Certaines stratégies utilisées pour améliorer la qualité de la génération de données comprennent:
- Comparaison des variantes génétiques identifiées entre le séquençage et le génotypage par matrice (si possible)
- Comparaison des SNP codants à un individu séquencé par le génome entier avec le trouble
- Comparaison des SNP de codage avec le séquençage Sanger des individus HapMap
Créer appels de variante , les programmes d’analyse identifient les variantes à partir des données de séquence pour créer divers fichiers. Certains des fichiers courants sont les fichiers FASTQ, BAM / CRAM et VCF.
Vous pouvez en savoir plus sur l’ensemble de la procédure de séquençage de l’exome sur l’ensemble du séquençage de l’exome page wiki .
Qui devrait se faire tester?
le Collège américain de génétique médicale et de génomique dispose de politiques concernant le moment où les méthodes de séquençage doivent être utilisées, les résultats qui peuvent survenir et ce que les résultats pourraient indiquer.
Plus précisément, ils recommandent que les tests génétiques soient envisagés pour certaines personnes, notamment:
- Les données de phénotype ou d’histoire familiale impliquent fortement une étiologie génétique, mais le phénotype ne correspond pas à un trouble spécifique pour lequel un test génétique ciblant un gène spécifique est disponible sur une base clinique
- Un patient présente un trouble génétique défini qui démontre un degré élevé d’hétérogénéité génétique, faisant de l’analyse WES ou WGS de plusieurs gènes simultanément une approche plus pratique
- Un patient présente un trouble génétique probable, mais les tests génétiques spécifiques disponibles pour ce phénotype n’ont pas permis de poser un diagnostic
- Un fœtus avec un trouble génétique probable dans lequel des tests génétiques spécifiques
Avantages du séquençage de l’exome entier
Technologie de séquençage d’exome entière, comme les autres Méthodes de test ADN , vise à utiliser le génome pour aider à diagnostiquer et guider les traitements des troubles mendéliens. Il fournit également aux utilisateurs des informations qui peuvent les aider à informer les membres de leur famille de leur risque.
Cette méthode présente plusieurs avantages par rapport à l’analyse d’un seul gène, l’une des méthodes les plus courantes de séquençage d’ADN. Premièrement, le séquençage complet de l’exome permet d’identifier un large éventail de mutations dans des gènes candidats qui ne sont pas testés. De cette manière, un utilisateur peut identifier les gènes de maladies dont il ne soupçonnait peut-être pas qu’ils étaient à risque. Deuxièmement, en clinique, le séquençage complet de l’exome permet d’identifier les cas où des mutations de différents gènes contribuent aux différents phénotypes chez le même patient.
Le séquençage de l’exome entier peut également être utilisé dans la recherche scientifique pour identifier des maladies rares héréditaires non détectables par les microréseaux. Par exemple, les variants d’allèles de plusieurs troubles sévères sont connus pour être localisés sur les exons mais sont également extrêmement rares dans la population. Ainsi, les tests de génotypage standard qui nécessitent de grandes bases de données d’allèles pour être efficaces manquent souvent ces variantes. Plusieurs études de cas soulignent l’utilité de toute l’approche de séquençage de l’exome. En 2009, un groupe de scientifiques utilisé le séquençage de l’exome entier pour identifier la mutation qui cause le syndrome de Bartter et la diarrhée congénitale au chlorure.
En 2011, Ambry Genetics était le première entreprise pour offrir un service de séquençage d’exome pour le diagnostic clinique des maladies. La société affirme que les résultats du séquençage de l’exome leur permettront de diagnostiquer des maladies pour lesquelles les approches diagnostiques traditionnelles ne sont pas applicables. Vous pouvez en savoir plus sur l’historique et les résultats de Génétique Ambry dans notre revue de produit!

Limitations du séquençage de l’exome entier
Séquençage d’exome entier et séquençage du génome entier : Ces deux procédures sont des technologies de séquençage de nouvelle génération. Bien que le séquençage de l’exome entier démontre certains avantages par rapport au génotypage basé sur des puces à ADN pour l’analyse clinique du risque de maladie, il présente également des limites. La différence la plus importante entre le séquençage de l’exome entier et du génome entier est la quantité d’ADN séquencée. Étant donné que le séquençage de l’exome entier décode seulement 1% du génome, il ne parvient pas à analyser les 99% restants, y compris les régions structurelles et non codantes. D’un autre côté, le séquençage du génome entier révèle la totalité de votre ADN qui comprend les 3 milliards de paires de bases (seules 40 millions de paires de bases sont couvertes par le séquençage complet de l’exome).
Combien coûte le séquençage complet de l’exome? Pendant un certain temps, le séquençage de l’exome entier a été considéré comme le type de séquençage d’ADN le plus bénéfique car il offrait une vue élargie des régions codant l’ADN par rapport au génotypage sur microréseau à un prix beaucoup plus bas que le séquençage du génome entier. Cependant, ce n’est plus le cas.
Le séquençage du génome entier peut être réalisé pour une fraction de son prix d’origine et fournir plus d’informations que le séquençage de l’exome entier. À Génomique des nébuleuses , vous pouvez faire séquencer l’intégralité de votre génome pour moins de 299 $. C’est comparable et même moins cher que de nombreux produits de séquençage d’exomes entiers à la maison.
Microarray chromosomique vs séquençage de l’exome entier vs séquençage du génome entier
| Génotypage de puces à ADN | Séquençage d’exome entier | Séquençage du génome entier | |
| Objectif | Identifier des variantes spécifiques | Décoder tous les gènes | Décodez tous les gènes ainsi que toutes les régions structurelles et non codantes |
| Débit | Faible débit | Haut débit | Haut débit |
| Nécessite l’identification préalable de la variante | Oui | Non | Non |
| Montant de la couverture | 0,5 million de positions (0,02% du génome) | 60 millions de postes (~ 1% du génome) | 6 milliards de positions (~ 100% du génome) |
| Coût | ~ 100 $ | ~ 300 $ | ~ 1 000 $ (mais Nebula Genomics le propose pour 299 $) |
Direct aux entreprises de séquençage d’exome complet
Diverses sociétés vendent des produits de séquençage d’exomes entiers. Certains de ces produits doivent être commandés auprès d’un fournisseur de soins de santé et peuvent être couverts par une assurance. Ces tests sont souvent utilisés pour diagnostiquer la maladie. Certaines entreprises offrent le service sous forme de tests à domicile directs aux consommateurs, qui sont analysés dans des laboratoires approuvés par CLIA et accrédités par CAP.Ainsi, même s’ils ne peuvent pas être utilisés comme outils de diagnostic, ils peuvent être présentés aux prestataires de soins de santé pour consultation.
23andMe a mené un programme pilote de séquençage d’exome complet qui a été annoncé en septembre 2011 et a été interrompu en 2012. Les consommateurs pouvaient obtenir des données exome au coût de 999 $. La société a fourni des données brutes et n’a pas proposé d’analyse.
- Génétique Ambry a été la première entreprise à offrir un service de séquençage d’exome pour le diagnostic clinique des maladies. Aujourd’hui, les clients peuvent commander des tests auprès de prestataires de soins de santé et la plupart des coûts sont couverts par une assurance.
- CircleDNA offre à domicile WES pour 189 $ – 629 $ et de nombreux tests comprennent le conseil génétique. Cette société collabore également avec DNAFit .
- Dante Labs ‘ WES commence à 849 $, à un prix beaucoup plus élevé que ses concurrents. Cette société propose également le séquençage du génome entier à partir de 599 $.
- Génomes complets offre WES à partir de 525 $.
- GeneDx a commencé à offrir WES en 2012. Leurs prix sont variables.
- HelixDNA offre WES à un prix raisonnable de 145 $ plus 299 $ supplémentaires pour une application utile.
- Tous les deux Invitae WES et Myriad Genetics proposer des tests de diagnostic. Ces tests, bien que coûteux, peuvent être couverts par une assurance.
- Tellmegen examine certaines régions de l’exome connues pour être liées à la maladie et à la santé. Les tests coûtent 235 $.
Vous pouvez en lire beaucoup plus avis de l’entreprise sur notre blog et consultez notre guide complet du meilleur kit de test ADN et autres tests à domicile .
